やや場違い感が否めないが、今週、CBI学会(計算情報化学生物学会)の2016年大会でポスター発表を行ってきた。
発表タイトルは『Efficient Similarity Search Using Multiple Reference Molecules on PG-Strom architecture』というもので、要は、創薬分野で新薬の候補となる化合物を類似度サーチする際に、PG-Stromを用いて高速かつIn-Databaseで行ってしまおうという試みである。
前提となる利用シーンは、例えば次のようなシチュエーションである。
ある疾患には特定のたんぱく質が作用する事が分かっており、また、世界各地で発表される研究論文の中には『このたんぱく質には〇〇〇という化学物質が活性を持っている』というような事が記載されている。こういった組合せは別に1:1という訳ではなく、別の研究論文には化合物△△△が、別の論文には□□□が、程度の多寡はあれ活性を持っていたという事が書かれている訳である。
研究者は、このような研究論文からデータを引っ張ってきて、『タンパク質Xに対して活性を持つ化学物質の組』というのを作る。これを便宜上、Query Chemical Compounds (Q)と称する事にする。
一方、薬剤の候補となる化合物の探索空間としては概ね1000万件程度が知られている。これを便宜上、Database Chemical Compounds (D)と称する事にする。これらがどのような化学的特徴を持つのかという事は分かっているものの、目的となるたんぱく質に活性を示すのか否かは個別に調べてみる必要がある。但し、調べるといってもそれなりに費用・時間を要するため、適切なものを効率的に絞り込まねばならない。

ここで使用するのが類似度探索(Similarity Search)である。既に学術論文等で目的のたんぱく質に活性を示す事が報告されている化学物質に"近い"化合物を、1000万件の探索空間から絞り込む事ができれば、関連しそうな化合物に一気に網をかける事ができる。
選択の基準となるのが、化合物同士の"近さ"であるが、幾つかの方法が考えられる。中心点距離や平均値を用いる方法もあるが、今回はk-NN(k-Nearest Neighbor)法を用いた。
これは、Q化合物群とあるD化合物間の距離(類似度スコア)を全て計算した上で、そのうち最も近傍にあるk個の距離の平均値を代表距離とするものである。
この時、類似度スコアの計算量としては概ねO(QxD)のオーダーとなる。
つまり、Q化合物群が1000個、D化合物群が1000万個である場合には、100億通りの組合せに対して類似度スコアを計算する必要があり、そこそこしんどい計算になる。
今回の研究で使用したアプローチは、Q化合物群、D化合物群を共にPostgreSQLのテーブルに格納し、ここから読み出したデータをPL/CUDA関数の引数として与え、この関数の内部で①類似度スコアの計算、②並び替え(ソート)、③上位k件の平均値導出、までを並列に実行してやろうというものである。
PL/CUDAというのはPG-Stromが提供するPostgreSQLのProcedural Languageの一つで、SQL関数の定義部分にCUDAコードを直接記述する事ができる。実行時には、このコードブロックがGPU Kernel関数のテンプレートの中にスポっと挿入され、PG-Stromのインフラを使ってコードをビルド、GPUでの並列実行を行う。
関数の引数や返り値は、本来はRAM<-->GPU間で転送する必要があるが、この辺はSQL関数の定義から機械的に生成できる部分であるので、PG-Stromが全て面倒をみてくれる。今回は、Matrixに相当するデータ構造としてPostgreSQLの2D配列データ構造を使用してデータの受け渡しを行った。
そして、ひとたびk-NN法類似度サーチのロジックをPL/CUDA関数に組み込めば、あとは見慣れたSQLのパズルである。
例えば、以下のようなクエリを用いてfinger_print_queryテーブルから読み込んだQ化合物群、finger_print_10m_matrixテーブルから読み込んだD化合物群を、PG-Stromの提供するarray_matrix()関数を用いて2D配列データに変換し、PL/CUDA関数の引数として与えている。
PL/CUDA関数の返り値もまた2D配列であるが、これも同様にPG-Stromの提供するmatrix_unnest()関数により、一般的なN行のレコードへと戻している。
PREPARE knn_sim_rand_10m_gpu_v2(int) -- arg1:@k-value
AS
SELECT float4_as_int4(key_id) key_id, similarity
FROM matrix_unnest((SELECT rbind(knn_gpu_similarity($1,Q.matrix,D.matrix))
FROM (SELECT cbind(array_matrix(id),
array_matrix(bitmap)) matrix
FROM finger_print_query
LIMIT 99999) Q,
(SELECT matrix
FROM finger_print_10m_matrix) D))
AS sim(key_id real, similarity real)
ORDER BY similarity DESC
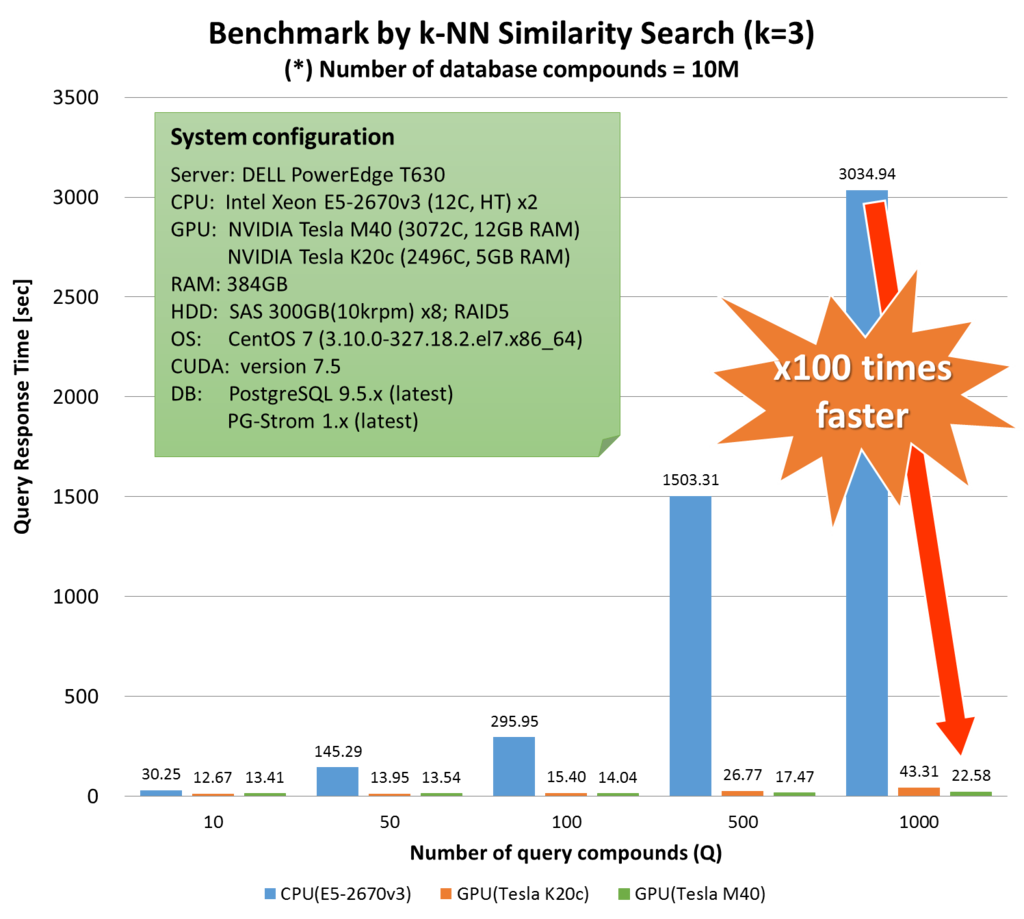
LIMIT 1000;ではこれで性能はどれ位出るのか?手元の環境で計測してみたところ、同じロジックをCPUで実装((knn_gpu_similarity()と等価なknn_cpu_similarity()関数をCで実装))して実行した場合と比べ、問題サイズが大きい場合では130倍を超える応答時間の改善を確認できた。
| Q-size | CPU(E5-2670v3) | GPU(Tesla K20) | GPU(Tesla M40) | 高速化率(CPU vs M40) |
|---|---|---|---|---|
| 10 | 30.25s | 13.23s | 13.41s | x2.3 |
| 50 | 145.29s | 14.49s | 13.54s | x10.7 |
| 100 | 295.95s | 15.99s | 14.04s | x21.1 |
| 500 | 1503.31s | 27.11s | 17.47s | x86.1 |
| 1000 | 3034.94s | 43.88s | 22.58s | x134.4 |
In-Databaseでのデータ解析処理でここまで応答時間が変わってくると、計算の運用に関わる従来の前提も色々と変わってくることになる。
一つ考えられるシナリオとしては、計算性能を担保するために外部のアプリケーションを使用してデータ解析を行っていたような利用シーンで、In-Database処理が十分な計算性能を出すようになった結果、データをエクスポートする必要がなくなるという事。これは、Hadoop方面の人が言うように『データのある場所で計算を行う』アプローチであり、最近の技術トレンドと合致している。
そしてもう一つの副次的な効果として、PL/CUDA関数の結果がSQLのデータ構造として返却されるという事は、データ解析ロジックの中で最もヘビーな計算処理を終えた後のデータに対して、JOIN、Window関数、ORDER BY、GROUP BYといったSQLの構文を使って非常にフレキシブルなデータの加工・後処理が可能になるという事である。
これらは、研究者がデータを多面的・多角的に眺めて新たな知見を得る事に役立つが、従来50分を要していた処理が22秒まで短縮された事と併せ、研究に不可欠なTRY&ERRORに適した環境を提供してくれることだろう。